Chapitre 5 Manipuler des données avec data.table
5.1 Tâches concernées et recommandations
L’utilisateur souhaite manipuler des données stucturées sous forme de data.frame (sélectionner des variables, sélectionner des observations, créer des variables, joindre des tables).
- Pour des tables de données de taille petite et moyenne (inférieure à 1 Go ou moins d’un million d’observations), il est recommandé d’utiliser le package
dplyr; - Pour des tables de données de grande taille (plus de 1 Go ou plus d’un million d’observations), il est recommandé d’utiliser le package
data.tablequi fait l’objet de la présente fiche.
L’utilisation du package data.table peut paraître plus déroutante pour les débutants que l’utilisation de dplyr. Toutefois, l’apprentissage de data.table est particulièrement recommandé si vous avez l’intention d’utiliser R avec des données volumineuses car data.table est beaucoup plus rapide et puissant que dplyr. Des remarques et conseils sont présents dans cette fiche pour vous aider à vous familiariser avec la syntaxe de data.table.
5.2 Présentation de data.table
Ne pas oublier de charger le package avec library(data.table).
5.2.1 Principes structurants
Le package data.table propose une version améliorée du data.frame de base : le data.table. La principale différence visible est que la visualisation d’un objet data.table est meilleure que celle d’un data.frame standard : le data.table indique automatiquement le type des variables (sous le nom de variable), et donne le nombre total d’observations de la table.
dt <- data.table(x = c("A", "B", "C"),
y = 1:12,
z = 3:6)
dt## x y z
## 1: A 1 3
## 2: B 2 4
## 3: C 3 5
## 4: A 4 6
## 5: B 5 3
## 6: C 6 4
## 7: A 7 5
## 8: B 8 6
## 9: C 9 3
## 10: A 10 4
## 11: B 11 5
## 12: C 12 6La fonction fondamentale de data.table est l’opérateur [...] (crochets). Lorsqu’on les applique à un objet data.frame de base, les crochets df[...] servent uniquement à sélectionner des lignes ou des colonnes. Dans un data.table, les crochets dt[...] permettent de faire beaucoup plus de choses (quasiment tout, en pratique). En fait, les instructions à l’intérieur des crochets peuvent être envisagées comme des requêtes SQL mises en forme différemment.
La forme générale de l’opérateur [...] est la suivante : DT[i, j, by]. Cette grammaire peut se lire comme ceci : "on part du data.table DT, on sélectionne certaines lignes avec i, puis on calcule j pour chaque groupe défini par by. Si on fait un parallèle avec SQL, i correspond au WHERE, j au SELECT et by au GROUP BY. La fonction [...] présente deux grands avantages :
- Il n’est pas nécessaire d’utiliser le préfixe
DT$pour se référer aux variables à l’intérieur de[...]; - Le code est très concis, ce qui aide à le rendre lisible.
Voici un exemple simple. A partir des données générées ci-dessus, on veut calculer la moyenne de y par groupe défini par x, uniquement sur les observations pour lesquelles x est supérieur à 3. Voici comment on peut réaliser cette opération avec Base R, dplyr et data.table. Vous pouvez juger vous-même de la concision du code.
5.2.2 Quelles fonctions peut-on utiliser avec un data.table ?
Les data.tables sont simplement des data.frames particuliers, donc on peut normalement leur appliquer toutes les méthodes valables pour les data.frames. En particulier, on peut utiliser avec data.table toutes les fonctions des packages habituellement associés à dplyr : stringr pour le maniement de chaînes de caractères, lubridate pour les colonnes temporelles, forcats pour les colonnes de type factor, etc. Toutefois, il est utile de vérifier que le package data.table ne propose pas déjà une fonction adaptée. Par exemple, plutôt que d’utiliser la fonction str_split_fixed() du package stringr pour séparer une colonne en fonction d’un caractère, on utilisera tstrsplit() de data.table.
5.2.3 Enchaîner les opérations en data.table
5.2.3.1 Le principe est simple…
Il est facile d’enchaîner des opérations avec data.table : il suffit d’accoler les opérateurs []. Votre code data.table prendra alors la forme suivante : dt[opération 1][opération 2][opération 3][...]. Voici un exemple simple, dans lequel on calcule la moyenne d’une variable par groupe, puis on trie la table.
## x moyenne
## 1: A 5.5
## 2: B 6.5
## 3: C 7.55.2.3.2 … mais il faut que le code reste lisible…
Le problème avec l’enchaînement d’opérations multiples est qu’on aboutit rapidement à des lignes de codes extrèmement longues. C’est pourquoi il est préférable de revenir régulièrement à la ligne, de façon à garder un code qui reste lisible. Il y a évidemment plusieurs façons d’organiser le code. La seule obligation est que le crochet qui commence une nouvelle opération doit être accolé au crochet qui termine l’opération précédente (...][...). Voici deux organisations possibles, à vous de choisir celle qui vous paraît la plus claire et la plus adaptée à votre travail.
La première organisation enchaîne toutes les opérations en une seule fois :
resultat <-
dt[i = ...,
j = ...,
by = ...
][i = ...,
j = ...,
by = ...
]La seconde organisation sépare les opérations en utilisant une table intermédiaire nommée resultat :
resultat <- dt[i = ...,
j = ...,
by = ...
]
resultat <- resultat[i = ...,
j = ...,
by = ...
]Comme indiqué précédemment, i, j et by ne sont pas forcément présents dans toutes les étapes. Voici ce que cette organisation du code donne sur un exemple légèrement plus complexe que le précédent :
dt[ , total := y + z]
resultat <- dt[ ,
.(moyenne = mean(total, na.rm = TRUE)),
by = x
][order(moyenne)]
resultat## x moyenne
## 1: A 10
## 2: B 11
## 3: C 125.2.3.3 … car on peut facilement faire des erreurs
L’enchaînement des opérations en data.table est puissant, mais peut aboutir à des résultats non désirés si on ne fait pas attention. Les exemples de ce paragraphe utilisent la fonction := ; si vous ne la connaissez pas encore, il est fortement conseillé de lire la section La fonction d’assignation par référence (ou :=) avant de poursuivre la lecture.
Voici deux exemples d’opérations enchaînées en data.table dont les codes sont très similaires et qui aboutissent à des résultats très différents. Le premier exemple ne conserve qu’une partie de la table dt puis crée une variable, tandis que le second crée une variable avec une valeur non manquante pour une partie de la table uniquement.
| Exemple 1 | Exemple 2 ``` | |
|---|---|---|
| Code |
|
|
| Signification |
Partir de dt, conserver uniquement les observations pour lesquelles x > 3, et créer une nouvelle variable newvar qui vaut 1 partout
|
Partir de dt, créer une nouvelle variable newvar qui vaut 1 pour les observations pour lesquelles x > 3 et NA ailleurs
|
5.3 Manipuler des tables de données avec data.table
Nous allons illustrer les fonctions de manipulation de données de data.table avec les jeux de données du package doremifasolData.
5.3.1 Mettre des données dans un data.table
Il y a principalement deux méthodes pour mettre des données sous forme d’un data.table :
- la fonction
fread()importe un fichier plat comme les.csv(voir la fiche Importer des fichiers plats (.csv,.tsv,.txt)) ; - Les fonctions
setDT()etas.data.table()convertissent undata.frameendata.table.
Dans la suite de cette section, on va illustrer les opérations de base en data.table avec la base permanente des équipements (table bpe_ens_2018), qu’on transforme en data.table.
# Charger la base permanente des équipements
bpe_ens_2018 <- bpe_ens_2018
# Convertir ce data.frame en data.table
bpe_ens_2018_dt <- as.data.table(bpe_ens_2018)
5.3.2 Manipuler une seule table avec data.table
5.3.2.1 Sélectionner des lignes
On peut sélectionner des lignes dans un data.table avec dt[i]. Voici un exemple de code qui sélectionne les magasins de chaussures (TYPEQU == "B304") dans le premier arrondissement de Paris (DEPCOM == "75101") dans la table bpe_ens_2018_dt :
selection <- bpe_ens_2018_dt[DEPCOM == "75101" & TYPEQU == "B304"]Voici une remarque très importante sur le fonctionnement de data.table : lorsqu’on souhaite conserver toutes les lignes d’un data.table, il faut laisser vide l’emplacement pour i, sans oublier la virgule. Par exemple, pour connaître le nombre de lignes de iris_dt, on écrit : iris_dt[ , .N]. Notez bien l’emplacement vide et la virgule après [.
5.3.2.2 Sélectionner des colonnes
On peut sélectionner des colonnes dans un data.table et renvoyer un data.table de plusieurs façons.
- La première consiste à indiquer les colonnes à conserver sous forme de liste. La notation
.()est un alias pourlist()qui est pratique et concis dans un codedata.table. Le code suivant sélectionne le code commune, le type d’équipement et le nombre d’équipement dans la base permanente des équipements :
bpe_ens_2018_dt[ , list(DEPCOM, TYPEQU, NB_EQUIP)]
bpe_ens_2018_dt[ , .(DEPCOM, TYPEQU, NB_EQUIP)]- La seconde méthode consiste à utiliser un mot-clé de
data.table,.SDqui signifieSubset of Data. On indique les colonnes qui seront aliasées par.SDavec la dimension.SDcols.
bpe_ens_2018_dt[ , .SD, .SDcols = c("DEPCOM", "TYPEQU", "NB_EQUIP")]La seconde méthode peut vous sembler inutilement complexe. C’est vrai dans l’exemple donné ci-dessus, mais les fonctions .SD et .SDcols s’avèrent très puissantes dans un grand nombre de situations (notamment quand on veut programmer des fonctions qui font appel à data.table).
5.3.2.3 Trier un data.table
On peut trier un data.table avec la fonction order(). Le code suivant trie la BPE selon le code commune et le type d’équipement.
bpe_ens_2018_dt[order(DEPCOM, TYPEQU)]## REG DEP DEPCOM DCIRIS AN TYPEQU NB_EQUIP
## 1: 84 01 01001 01001 2018 A401 2
## 2: 84 01 01001 01001 2018 A404 4
## 3: 84 01 01001 01001 2018 A504 1
## 4: 84 01 01001 01001 2018 A507 1
## 5: 84 01 01001 01001 2018 B203 1
## ---
## 1035560: 06 976 97617 97617 2018 F111 4
## 1035561: 06 976 97617 97617 2018 F113 4
## 1035562: 06 976 97617 97617 2018 F114 1
## 1035563: 06 976 97617 97617 2018 F120 1
## 1035564: 06 976 97617 97617 2018 F121 3Il suffit d’ajouter un signe - devant une variable pour trier par ordre décroissant. Le code suivant trie la BPE par code commune croissant et type d’équipement décroissant.
bpe_ens_2018_dt[order(DEPCOM, -TYPEQU)]5.3.2.4 Calculer des statistiques
La méthode pour sélectionner des colonnes est également valable pour calculer des statistiques, car data.table accepte les expressions dans j. Le code suivant calcule le nombre total d’équipements dans la BPE (sum(NB_EQUIP, na.rm = TRUE) :
bpe_ens_2018_dt[ , .(sum(NB_EQUIP, na.rm = TRUE))]## V1
## 1: 2504782Il est possible de calculer plusieurs statistiques à la fois, et de donner des noms aux variables ; il suffit de séparer les formules par une virgule. Le code suivant calcule le nombre total d’équipements dans la BPE (sum(NB_EQUIP, na.rm = TRUE)), et le nombre total de boulangeries (sum(NB_EQUIP * (TYPEQU == "B203"), na.rm = TRUE)).
bpe_ens_2018_dt[ ,
.(NB_EQUIP_TOT = sum(NB_EQUIP, na.rm = TRUE),
NB_BOULANG_TOT = sum(NB_EQUIP * (TYPEQU == "B203"), na.rm = TRUE))]## NB_EQUIP_TOT NB_BOULANG_TOT
## 1: 2504782 48568On peut évidemment combiner i et j pour calculer des statistiques sur un sous-ensemble d’observations. Dans l’exemple suivant, on sélectionne les boulangeries avec i ((TYPEQU == "B203")), et on calcule le nombre total d’équipements avec j (sum(NB_EQUIP, na.rm = TRUE)).
bpe_ens_2018_dt[TYPEQU == "B203", .(NB_BOULANG_TOT = sum(NB_EQUIP, na.rm = TRUE))]## NB_BOULANG_TOT
## 1: 48568
5.3.2.5 Les fonctions statistiques utiles de data.table
Vous pouvez utiliser toutes les fonctions statistiques de R avec data.table. Le package data.table propose par ailleurs des fonctions optimisées qui peuvent vous être utiles. En voici quelques-unes :
| Fonction | Opération | Exemple |
|---|---|---|
.N |
Nombre d’observations | dt[ , .N, by = 'group_var'] |
uniqueN() |
Nombre d’observations uniques de la variable x
|
dt[ , uniqueN(x), by = 'group_var'] |
%in% |
Nombre dans la liste | dt[x %in% 1:5] |
%chin% |
Chaîne de caractères dans la liste | dt[x %chin% c("a", "b")] |
%between% |
Valeur entre deux nombres | dt[x %between% c(5,13)] |
%like% |
Reconnaissance d’une chaîne de caractères | dt[departement %like% "^Haute"] |
5.3.2.6 Opérations par groupe
Toutes les opérations précédentes peuvent être réalisées par groupe. il suffit d’ajouter le nom des variables de groupe dans by (c’est l’équivalent du group_by() du package dplyr). Lorsqu’il y a plusieurs variables de groupe, on peut écrire l’argument by de deux façons :
- soit
by = c("var1", "var2", "var3")(attention aux guillemets) ; - soit
by = .(var1, var2, var3)(attention à la notation.()).
Le code suivant groupe les données de la BPE par département (by = .(DEP)) puis calcule le nombre total d’équipements (sum(NB_EQUIP, na.rm = TRUE)) et le nombre total de boulangeries (sum(NB_EQUIP * (TYPEQU == "B203"), na.rm = TRUE)).
bpe_ens_2018_dt[ ,
.(NB_EQUIP_TOT = sum(NB_EQUIP, na.rm = TRUE),
NB_BOULANG_TOT = sum(NB_EQUIP * (TYPEQU == "B203"), na.rm = TRUE)),
by = .(DEP)]## DEP NB_EQUIP_TOT NB_BOULANG_TOT
## 1: 01 21394 401
## 2: 02 15534 339
## 3: 03 12216 299
## 4: 04 8901 185
## 5: 05 8785 175
## ---
## 97: 971 23939 516
## 98: 972 19068 370
## 99: 973 7852 98
## 100: 974 30767 646
## 101: 976 7353 101L’argument by fonctionne également avec l’opérateur :=. Vous pouvez en apprendre davantage sur l’usage de cet opérateur dans la partie La fonction d’assignation par référence (ou :=).
5.3.3 Joindre des tables avec data.table
Pour joindre des données, data.table propose une fonction merge() plus rapide que la fonction de base. La syntaxe générale est z <- merge(x, y, [options]). Voici une liste des principales options (les autres options sont visibles avec ?data.table::merge) :
| Option | Signification |
|---|---|
by = var_jointure |
Joindre sur la variable var_jointure (présente dans x et dans y) |
by.x = "identx", by.y = "identy" |
Joindre sur la condition identx == identy
|
all.x = TRUE |
Left join (garder toutes les lignes de x) |
all.y = TRUE |
Right join (garder toutes les lignes de y) |
all = TRUE |
Full join (garder toutes les lignes de x et de y) |
Enfin, il est possible de réalier des jointures plus sophistiquées avec data.table. Ces méthodes sont présentées dans la vignette sur le sujet.
5.3.4 Indexer une table avec data.table
L’indexation est une fonctionalité très puissante pour accélérer les opérations sur les lignes (filtres, jointures, etc.) en data.table. Pour indexer une table il faut déclarer les variables faisant office de clé (appelées key). C’est possible de la manière suivante : setkey(dt, a) ou setkeyv(dt, "a"). Le data.table sera réordonné en fonction de cette variable et l’algorithme de recherche sur les lignes sera ainsi beaucoup plus efficace. Lorsqu’il y a plusieurs variables-clé, on écrit setkey(dt, a, b) ou setkeyv(dt, c("a","b")).
Pour savoir si un data.table est déjà indexé, on peut exécuter la commande key(dt) qui renvoie le nom des clés s’il y en a, et NULL sinon.
L’exécution de la fonction data.table::setkey() peut prendre un peu de temps (parfois quelques minutes sur une table de plus de 10 millions de lignes), car data.table trie toute la table en fonction des variables-clé. Toutefois, c’est une étape vraiment utile car elle accélère considérablement les opérations ultérieures sur les lignes. Il est vivement recommandé de l’utiliser si une ou plusieurs variables vont régulièrement servir à filtrer ou combiner des données. Pour aller plus loin, voir cette vignette.
5.3.5 Réorganiser les données en data.table
Le package data.table permet de réorganiser facilement une table de données avec les fonctions dcast() et melt(). La fonction melt() réorganise les donnée dans un format long. La fonction dcast() réorganise les donnée dans un format wide.
melt() |
dcast() |
|---|---|
Réorganiser les donnée dans un format long
|
Réorganise les donnée dans un format wide
|
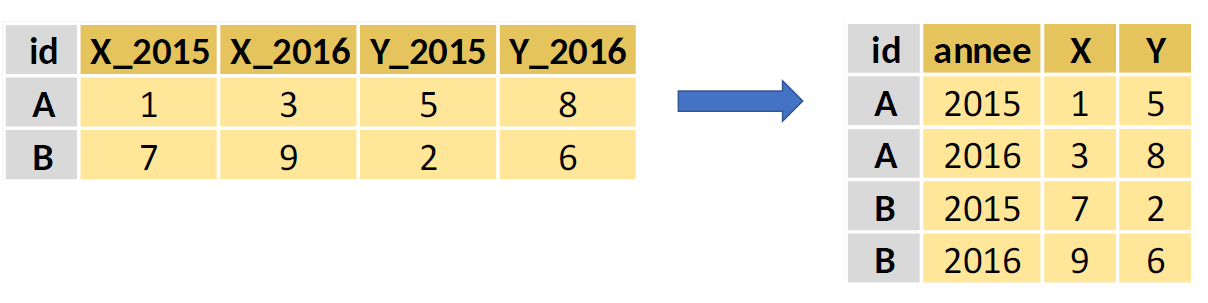 |
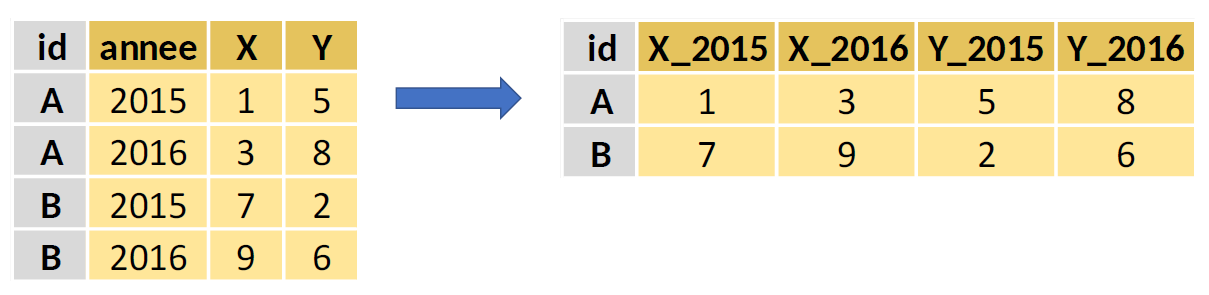 |
5.3.5.1 melt : transformer des colonnes en lignes
La fonction melt() réorganise les donnée dans un format long. Elle prend les arguments suivants :
-
data: les données ; -
id.vars: les variables qui identifient les lignes de table d’arrivée ; elles restent inchangées lors de l’utilisation demelt(); -
measure.vars: les variables qui sont transposées ; -
variable.name: le nom de la nouvelle colonne qui contient le nom des variables transposées ; -
value.name: le nom de la nouvelle colonne qui contient la valeur des variables transposées.
Pour illustrer l’usage de cette fonction, nous allons utiliser les données du répertoire Filosofi 2016 agrégées au niveau des EPCI (table filosofi_epci_2016), et disponibles dans le package doremifasolData. On convertit cette table en data.table et on conserve uniquement certaines variables.
# Charger la table de Filosofi
filosofi_epci_2016 <- filosofi_epci_2016
# Convertir la table en data.table
filosofi_epci_2016_dt <- as.data.table(filosofi_epci_2016)
# Sélectionner des colonnes
filosofi_epci_2016_dt <-
filosofi_epci_2016_dt[, .(CODGEO, TP6016, TP60AGE116, TP60AGE216,
TP60AGE316, TP60AGE416, TP60AGE516, TP60AGE616)]Nous allons restructurer cette table pour obtenir une nouvelle table, avec une observation par EPCI et par tranche d’âge. Voici le code qui permet d’obtenir cette table : on indique dans measure.vars le nom des colonnes qui seront transposées, le nom des colonnes transposées sera indiqué dans la nouvelle colonne “tranche_age” (variable.name = "tranche_age") et les valeurs des colonnes transposées seront stockées dans la colonne “taux_pauvrete” (value.name = "taux_pauvrete").
donnees_pauvrete_long <-
melt(data = filosofi_epci_2016_dt,
c("CODGEO"),
measure.vars = c("TP6016", "TP60AGE116", "TP60AGE216",
"TP60AGE316", "TP60AGE416", "TP60AGE516", "TP60AGE616"),
variable.name = "tranche_age",
value.name = "taux_pauvrete"
)
donnees_pauvrete_long## CODGEO tranche_age taux_pauvrete
## 1: 200000172 TP6016 8.8
## 2: 200000438 TP6016 8.0
## 3: 200000545 TP6016 23.7
## 4: 200000628 TP6016 20.1
## 5: 200000800 TP6016 11.4
## ---
## 8704: 249740077 TP60AGE616 44.3
## 8705: 249740085 TP60AGE616 41.5
## 8706: 249740093 TP60AGE616 43.4
## 8707: 249740101 TP60AGE616 39.8
## 8708: 249740119 TP60AGE616 31.7Il est recommandé de travailler avec des données en format long plutôt qu’en format wide, notamment lorsque vous voulez faire des graphiques. En effet, le package de visualisation graphique ggplot2 est optimisé pour manipuler des données en format long (voir la fiche [Faire des graphiques avec ggplot2]). Ce conseil est particulièrement important si vous voulez représenter un graphique avec des groupes : il est préférable que les groupes soient empilés (format long) plutôt que juxtaposés (format wide), car le code est plus rapide et facile à écrire.
5.3.5.2 dcast : transformer des lignes en colonnes
La fonction dcast() réorganise les donnée dans un format large. Elle prend les arguments suivants :
-
data: les données ; -
formula: une formule de la formevar_ligne ~ var_colonnequi définit la structure de la nouvelle table ;- s’il y a plusieurs variables, la formule prend la forme
var1 + var2 ~ var3; -
dcast()conserve une ligne par valeur de la partie gauche, et crée (au moins) une colonne par valeur de la partie droite ;
- s’il y a plusieurs variables, la formule prend la forme
-
fun.aggregate: une liste contenant la ou les fonction(s) utilisées pour agréger les données le cas échéant ; exemple :list(mean, sum, sd); -
value.var: un vecteur contenant le nom de la ou des colonne(s) dont les valeurs vont être transposées ; exemple :c("var1", "var2").
Dans l’exemple qui suit, on réorganise la table bpe_ens_2018_dt de façon à obtenir une table qui contient une ligne par type d’équipement et une colonne par région (TYPEQU ~ REG). Ces colonnes vont contenir la somme (fun.aggregate = sum) du nombre d’équipements (value.var = "NB_EQUIP").
bpe_ens_2018_wide <- dcast(bpe_ens_2018_dt,
TYPEQU ~ REG,
value.var = "NB_EQUIP",
fun.aggregate = sum)
head(bpe_ens_2018_wide)## TYPEQU 01 02 03 04 06 11 24 27 28 32 44 52 53 75 76 84 93 94
## 1: A101 2 2 1 7 0 191 28 23 54 127 80 15 20 66 55 69 34 3
## 2: A104 20 21 16 28 5 91 153 230 183 214 319 173 157 407 406 423 179 39
## 3: A105 1 1 1 1 1 2 2 2 2 2 4 1 1 5 3 4 1 1
## 4: A106 2 1 2 2 1 10 7 12 10 17 17 8 8 19 19 21 11 2
## 5: A107 2 1 1 4 1 60 9 19 15 26 30 11 12 28 26 34 23 2
## 6: A108 2 1 1 2 1 19 9 13 13 25 21 8 10 21 20 28 14 2Il est possible d’utiliser dcast() avec plusieurs variables à transposer et plusieurs fonctions pour transposer. Dans l’exemple qui suit, on obtient une ligne par type d’équipement, et une colonne par région et par fonction d’agrégation (mean et sum).
bpe_ens_2018_wide2 <- dcast(bpe_ens_2018_dt,
TYPEQU ~ REG,
value.var = "NB_EQUIP",
fun.aggregate = list(sum, mean))
bpe_ens_2018_wide2## TYPEQU NB_EQUIP_sum_01 NB_EQUIP_sum_02 NB_EQUIP_sum_03 NB_EQUIP_sum_04
## 1: A101 2 2 1 7
## 2: A104 20 21 16 28
## 3: A105 1 1 1 1
## 4: A106 2 1 2 2
## 5: A107 2 1 1 4
## ---
## 182: F306 1 1 2 7
## 183: G101 105 79 48 136
## 184: G102 49 49 29 112
## 185: G103 0 0 0 0
## 186: G104 110 104 46 98
## NB_EQUIP_sum_06 NB_EQUIP_sum_11 NB_EQUIP_sum_24 NB_EQUIP_sum_27
## 1: 0 191 28 23
## 2: 5 91 153 230
## 3: 1 2 2 2
## 4: 1 10 7 12
## 5: 1 60 9 19
## ---
## 182: 0 372 36 34
## 183: 20 3351 213 256
## 184: 11 2478 670 890
## 185: 0 96 238 330
## 186: 6 2993 293 339
## NB_EQUIP_sum_28 NB_EQUIP_sum_32 NB_EQUIP_sum_44 NB_EQUIP_sum_52
## 1: 54 127 80 15
## 2: 183 214 319 173
## 3: 2 2 4 1
## 4: 10 17 17 8
## 5: 15 26 30 11
## ---
## 182: 55 62 69 49
## 183: 272 495 593 376
## 184: 845 698 1318 762
## 185: 378 521 368 646
## 186: 401 354 533 330
## NB_EQUIP_sum_53 NB_EQUIP_sum_75 NB_EQUIP_sum_76 NB_EQUIP_sum_84
## 1: 20 66 55 69
## 2: 157 407 406 423
## 3: 1 5 3 4
## 4: 8 19 19 21
## 5: 12 28 26 34
## ---
## 182: 51 88 80 121
## 183: 345 720 786 1166
## 184: 943 1908 1982 2797
## 185: 751 1408 1437 1265
## 186: 374 926 932 1099
## NB_EQUIP_sum_93 NB_EQUIP_sum_94 NB_EQUIP_mean_01 NB_EQUIP_mean_02
## 1: 34 3 1.000000 1.000000
## 2: 179 39 1.000000 1.000000
## 3: 1 1 1.000000 1.000000
## 4: 11 2 1.000000 1.000000
## 5: 23 2 1.000000 1.000000
## ---
## 182: 75 5 1.000000 1.000000
## 183: 1016 120 2.282609 1.975000
## 184: 2111 438 2.450000 2.130435
## 185: 718 187 NaN NaN
## 186: 876 182 1.718750 1.575758
## NB_EQUIP_mean_03 NB_EQUIP_mean_04 NB_EQUIP_mean_06 NB_EQUIP_mean_11
## 1: 1.000000 1.000000 NaN 1.091429
## 2: 1.000000 1.000000 1.000000 1.000000
## 3: 1.000000 1.000000 1.000000 1.000000
## 4: 1.000000 1.000000 1.000000 1.000000
## 5: 1.000000 1.000000 1.000000 1.000000
## ---
## 182: 1.000000 1.000000 NaN 1.144615
## 183: 1.714286 1.837838 5.000000 2.080074
## 184: 1.318182 2.036364 1.833333 2.250681
## 185: NaN NaN NaN 1.103448
## 186: 1.533333 1.400000 1.500000 1.780488
## NB_EQUIP_mean_24 NB_EQUIP_mean_27 NB_EQUIP_mean_28 NB_EQUIP_mean_32
## 1: 1.037037 1.000000 1.018868 1.058333
## 2: 1.000000 1.004367 1.000000 1.014218
## 3: 1.000000 1.000000 1.000000 1.000000
## 4: 1.000000 1.000000 1.000000 1.000000
## 5: 1.000000 1.000000 1.000000 1.000000
## ---
## 182: 1.058824 1.096774 1.000000 1.033333
## 183: 1.601504 1.422222 1.511111 1.633663
## 184: 1.763158 1.666667 1.978923 1.681928
## 185: 1.048458 1.103679 1.330986 1.527859
## 186: 1.140078 1.232727 1.297735 1.156863
## NB_EQUIP_mean_44 NB_EQUIP_mean_52 NB_EQUIP_mean_53 NB_EQUIP_mean_75
## 1: 1.025641 1.000000 1.000000 1.157895
## 2: 1.009494 1.005814 1.000000 1.007426
## 3: 1.000000 1.000000 1.000000 1.000000
## 4: 1.000000 1.000000 1.000000 1.000000
## 5: 1.000000 1.000000 1.000000 1.000000
## ---
## 182: 1.095238 1.042553 1.085106 1.023256
## 183: 1.694286 1.748837 1.674757 1.578947
## 184: 1.734211 1.836145 2.063457 1.927273
## 185: 1.153605 2.044304 1.891688 1.733990
## 186: 1.230947 1.274131 1.307692 1.293296
## NB_EQUIP_mean_76 NB_EQUIP_mean_84 NB_EQUIP_mean_93 NB_EQUIP_mean_94
## 1: 1.145833 1.029851 1.000000 1.000000
## 2: 1.015000 1.011962 1.028736 1.083333
## 3: 1.000000 1.000000 1.000000 1.000000
## 4: 1.000000 1.000000 1.000000 1.000000
## 5: 1.000000 1.000000 1.000000 1.000000
## ---
## 182: 1.095890 1.080357 1.190476 1.000000
## 183: 1.526214 1.707174 1.785589 2.000000
## 184: 2.045408 2.109351 2.507126 3.369231
## 185: 1.600223 1.408686 1.681499 2.101124
## 186: 1.226316 1.345165 1.364486 1.857143La fonction
dcast()crée une colonne par valeur des variables utilisées dans la partie droite de la formule (trois colonnes pour les trois valeurs decyldans l’exemple qui précède). Il faut donc faire attention à ce que ces variables aient un nombre limité de valeurs, pour ne pas obtenir une table extrêmement large. On peut éventuellement discrétiser les variables continues, ou regrouper les modalités avant d’utiliserdcast().-
On peut obtenir des noms de colonnes peu significatifs lorsqu’on utilise
dcast()avec une fonction d’agrégation (01,02,03… dans l’exemple précédent). Il est conseillé de modifier légèrement la partie droite de la formule pour obtenir des noms plus significatifs. Voici un exemple où on ajoute le préfixeresultat_region:bpe_ens_2018_wide2 <- dcast(bpe_ens_2018_dt, TYPEQU ~ paste0("resultat_region",REG), value.var = "NB_EQUIP", fun.aggregate = sum) head(bpe_ens_2018_wide2)## TYPEQU resultat_region01 resultat_region02 resultat_region03 ## 1: A101 2 2 1 ## 2: A104 20 21 16 ## 3: A105 1 1 1 ## 4: A106 2 1 2 ## 5: A107 2 1 1 ## 6: A108 2 1 1 ## resultat_region04 resultat_region06 resultat_region11 resultat_region24 ## 1: 7 0 191 28 ## 2: 28 5 91 153 ## 3: 1 1 2 2 ## 4: 2 1 10 7 ## 5: 4 1 60 9 ## 6: 2 1 19 9 ## resultat_region27 resultat_region28 resultat_region32 resultat_region44 ## 1: 23 54 127 80 ## 2: 230 183 214 319 ## 3: 2 2 2 4 ## 4: 12 10 17 17 ## 5: 19 15 26 30 ## 6: 13 13 25 21 ## resultat_region52 resultat_region53 resultat_region75 resultat_region76 ## 1: 15 20 66 55 ## 2: 173 157 407 406 ## 3: 1 1 5 3 ## 4: 8 8 19 19 ## 5: 11 12 28 26 ## 6: 8 10 21 20 ## resultat_region84 resultat_region93 resultat_region94 ## 1: 69 34 3 ## 2: 423 179 39 ## 3: 4 1 1 ## 4: 21 11 2 ## 5: 34 23 2 ## 6: 28 14 2
Il est conseillé de bien réfléchir avant de restructurer des données en format wide, et de ne le faire que lorsque cela paraît indispensable. En effet, s’il est tentant de restructurer les données sous format wide car ce format peut paraître plus intuitif, il est généralement plus simple et plus rigoureux de traiter les données en format long. Ceci dit, il existe des situations dans lesquelles il est indiqué de restructurer les données en format wide. Voici deux exemples :
- produire un tableau synthétique de résultats, prêt à être diffusé, avec quelques colonnes donnant des indicateurs par catégorie (exemple : la table
filosofi_epci_2016du packagedoremifasolData) ; - produire une table avec une colonne par année, de façon à calculer facilement un taux d’évolution entre deux dates.
5.4 La fonction d’assignation par référence (ou :=)
Jusqu’à présent, mous avons manipulé un data.table existant, mais nous ne lui avons pas ajouté de nouvelles colonnes. Pour ce faire, nous allons utiliser la fonction := (qui s’appelle “assignation par référence” et qui peut également s’écrire `:=`()). Voici comment on crée une nouvelle colonne dans bpe_ens_2018_dt :
bpe_ens_2018_dt[ , nouvelle_colonne := NB_EQUIP * 10]
head(bpe_ens_2018_dt)## REG DEP DEPCOM DCIRIS AN TYPEQU NB_EQUIP nouvelle_colonne
## 1: 84 01 01001 01001 2018 A401 2 20
## 2: 84 01 01001 01001 2018 A404 4 40
## 3: 84 01 01001 01001 2018 A504 1 10
## 4: 84 01 01001 01001 2018 A507 1 10
## 5: 84 01 01001 01001 2018 B203 1 10
## 6: 84 01 01001 01001 2018 C104 1 10
5.4.1 La spécificité de data.table : la modification par référence
A première vue, on peut penser que la fonction := est l’équivalent de la fonction dplyr::mutate() dans la grammaire data.table. C’est vrai dans la mesure où elle permet de faire des choses similaires, mais il faut garder en tête que son fonctionnement est complètement différent de celui de dplyr::mutate(). En effet, la grande spécificité de data.table par rapport à dplyr est que l’utilisation de la fonction := modifie directement la table de données, car data.table fonctionne sur le principe de la modification par référence (voir ce lien
pour plus de détails). Cela signifie en pratique qu’il ne faut pas réassigner l’objet lorsqu’on modifie une de ses colonnes. C’est ce comportement qui permet à data.table d’être très rapide et très économe en mémoire vive, rendant son usage approprié pour des données volumineuses.
Pour créer une colonne, on écrit donc directement dt[ , nouvelle_colonne := une_formule] et non dt <- dt[ , nouvelle_colonne := une_formule]. Voici un exemple qui compare dplyr et data.table :
| Package | Code | Commentaire |
|---|---|---|
dplyr
|
|
Il faut utiliser une assignation (<-) pour modifier la table.
|
data.table
|
|
Il ne faut pas d’assignation pour modifier la table, qui est modifiée par référence. |
5.4.2 Les usages de :=
On peut se servir de la fonction := de multiples façons, et avec plusieurs notations.
5.4.2.1 Créer plusieurs variables à la fois
Voici comment créer plusieurs variables à la fois avec :=, en utilisant une notation vectorielle :
On peut faire exactement la même chose en utilisant la notation `:=`(). Voici le même exemple écrit avec `:=`().
bpe_ens_2018_dt[ , `:=`(nouvelle_colonne1 = NB_EQUIP * 2,
nouvelle_colonne2 = NB_EQUIP + 3)]Si vous utilisez la notation`:=`(), alors il faut utiliser uniquement = à l’intérieur des parenthèses pour créer ou modifier des variables, et non :=. Exemple :
dt[ , `:=`(var1 = "Hello", var2 = "world")]5.4.2.2 Supprimer une colonne
On peut facilement supprimer une colonne en lui assignant la valeur NULL (c’est hyper rapide !). Voici un exemple :
bpe_ens_2018_dt[ , NB_EQUIP := NULL]5.4.2.3 Faire un remplacement conditionnel
La fonction := peut être utilisée pour modifier une colonne pour certaines lignes seulement, en fonction d’une condition logique. C’est beaucoup plus efficace qu’un terme dplyr::if_else() ou dplyr::case_when(). Imaginons qu’on veuille créer une colonne EQUIP_HORS_CHAUSS égale au nombre d’équipements (NB_EQUIP) sauf pour les lignes correspondantes à des magasins de chaussures (TYPEQU == "B304") où elle vaut NA. Dans ce cas, le code dplyr serait :
bpe_ens_2018 %>%
dplyr::mutate(NB_EQUIP_HORS_CHAUSS = dplyr::case_when(
TYPEQU ~ "B304" == NA_real_,
TRUE ~ NB_EQUIP)
)et le code équivalent en data.table, nécessitant beaucoup moins de mémoire vive :
bpe_ens_2018_dt[ , NB_EQUIP_HORS_CHAUSS := NB_EQUIP
][TYPEQU == "B304", NB_EQUIP_HORS_CHAUSS := NA_real_]
5.4.3 Attention en utilisant :=
L’utilisation de la fonction := est déroutante lorsqu’on découvre data.table. Voici trois remarques qui vous feront gagner du temps :
-
Vous pouvez faire appel à d’autres fonctions à l’intérieur de la fonction
:=. Par exemple, si on veut mettre la variablenameen minuscules, on peut utiliser la fonctiontolower(). On écrit alors :dt[ , name_minuscule := tolower(name)] -
Lorsque l’on crée plusieurs variables avec la fonction
:=, elles sont créées en même temps. On ne peut donc pas faire appel dans une formule à une variable qu’on crée dans le même appel à la fonction:=. Par exemple, le code suivant ne fonctionne pas :bpe_ens_2018_dt[ , `:=`(nouvelle_colonne1 = NB_EQUIP * 2, nouvelle_colonne2 = nouvelle_colonne1 + 3)]En effet, au moment où la fonction
:=est exécutée, la colonnenouvelle_colonne1n’existe pas encore, donc la formulenouvelle_colonne2 = nouvelle_colonne1 + 3n’a pas encore de sens. Si vous créez des variables en chaîne, il faut décomposer l’opération en plusieurs étapes enchaînées. Voici le code qui permet de créer les deux colonnes à la suite :bpe_ens_2018_dt[ , `:=`(nouvelle_colonne1 := NB_EQUIP * 2 ][ , nouvelle_colonne2 := nouvelle_colonne1 + 3)] -
Un mauvais usage de la fonction
:=peut vous amener à écraser par erreur vos données. En effet, si vous exécuter par erreur la commandedt[ , ma_variable_importante := 0], vous écrasez la variablema_variable_importante. Vous devez alors recharger vos données… Il faut donc bien réfléchir à ce que vous voulez faire avant de remplacer ou modifier une variable existante avec la fonction:=. Si vous modifiez undata.tabledans une fonction, un filet de sécurité consiste à d’abord copier ledata.tableinitial et ainsi faire les modifications sur le nouvel objet, de la manière suivante :dt_copy <- data.table::copy(dt) dt_copy[, ma_variable_importante := "Nouvelle valeur"]
5.5 Programmer des fonctions avec data.table
Une des forces de data.table est qu’il est relativement simple d’utiliser ce package dans des fonctions. Pour illustrer l’usage des fonctions, nous allons utiliser la table filosofi_com_2016 disponible dans le package doremifasolData. Cette table donne des informations sur les revenus des ménages au niveau communal. Nous créons une variable donnant le numéro du département (departement) en extrayant les deux premiers caractères du code commune (CODGEO) avec la fonction str_sub du package stringr (vous pouvez consulter la fiche [Manipuler des données textuelles pour en apprendre davantage sur stringr]). Enfin, nous utilisons la fonction .SD pour sélectionner uniquement quelques variables.
# Charger la table de données et la transformer en data.table
filosofi_com_2016_dt <- as.data.table(doremifasolData::filosofi_com_2016)
# Créer une variable donnant le département
filosofi_com_2016_dt[, departement := stringr::str_sub(CODGEO, start = 1L, end = 2L)]
# Supprimer les départements d'outre-mer
filosofi_com_2016_dt <- filosofi_com_2016_dt[departement != "97"]
# Alléger la table en ne conservant que quelques variables
filosofi_com_2016_dt <-
filosofi_com_2016_dt[, .SD, .SDcols = c("departement", "CODGEO", "NBMENFISC16",
"NBPERSMENFISC16")]
5.5.1 Utiliser .SD et lapply
Le mot clé .SD (Subset of Data) permet d’appliquer la même opération sur plusieurs colonnes. Les colonnes auxquelles l’opération s’applique sont contrôlées par l’argument .SDcols (par défaut, toutes les colonnes sont traitées). Le mot clé .SD est régulièrement utilisé en conjonction avec la fonction lapply. Cette syntaxe, très puissante, permet également d’avoir des codes assez compacts, ce qui les rend plus lisible.
Un usage classique de ce duo lapply+.SD consiste à écrire des fonctions de statistiques descriptives. Par exemple, imaginons qu’on souhaite calculer la moyenne, l’écart-type et les quantiles (P25, P50 et P75) de nombreuses colonnes. On peut alors définir la fonction suivante :
mes_statistiques <-
function(x) return(c(mean(x, na.rm = TRUE),
sd(x, na.rm = TRUE),
quantile(x, probs = c(.25,.5,.75), na.rm = TRUE)))Voici comment on peut appliquer cette fonction aux colonnes NBMENFISC16 (nombre de ménages fiscaux) et NBPERSMENFISC16 (nombre de personnes dans les ménages fiscaux) de la table filosofi_com_2016_dt :
data_agregee <-
filosofi_com_2016_dt[ ,
lapply(.SD, mes_statistiques),
.SDcols = c("NBMENFISC16", "NBPERSMENFISC16")]
data_agregee[, 'stat' := c("moyenne","écart-type","P25","P50","P75")]
data_agregee## NBMENFISC16 NBPERSMENFISC16 stat
## 1: 916.3269 2097.18 moyenne
## 2: 7382.4628 15245.60 écart-type
## 3: 105.0000 250.50 P25
## 4: 218.0000 527.00 P50
## 5: 526.0000 1278.25 P75Il est également très simple d’effectuer des calculs par groupe avec la méthode lapply+.SD. On peut par facilement adapter le code précédent pour calculer des statistiques descriptives par département (variable departement).
5.5.2 Définir des fonctions modifiant un data.table
Il est très facile d’écrire avec data.table des fonctions génériques faisant appel à des noms de variables en arguments. Pour déclarer à data.table qu’un nom fait référence à une colonne, la manière la plus simple est d’utiliser la fonction get. Dans l’exemple suivant, on définit la fonction creation_var qui crée dans la table data une nouvelle variable (dont le nom est l’argument nouveau_nom) égale à une autre variable incrémentée (dont le nom est l’argument nom_variable) de 1. L’utilisation de la fonction get permet d’indiquer à data.table que la chaîne de caractères nom_variable désigne une colonne de la table data.
creation_var <- function(data, nom_variable, nouveau_nom){
data[, c(nouveau_nom) := get(nom_variable) + 1]
}
head(creation_var(filosofi_com_2016_dt,
nom_variable = "NBMENFISC16",
nouveau_nom = "nouvelle_variable"), 2)## departement CODGEO NBMENFISC16 NBPERSMENFISC16 nouvelle_variable
## 1: 01 01001 313 795.5 314
## 2: 01 01002 101 248.0 102c(nouveau_nom) permet de s’assurer que data.table crée une nouvelle colonne dont le nom est défini en argument (et qui ne s’appelle donc pas nouveau_nom).
Il est particulièrement complexe d’écrire avec dplyr des fonctions génériques faisant appel à des noms de variables en arguments : il est nécessaire de faire appel à plusieurs fonctionnalités de rlang pour être en mesure d’effectuer une opération équivalente. Voici pour information la version dplyr de l’exemple précédent :
modif_var <- function(data, nom_variable){
data %>%
dplyr::mutate(nouveau_nom = !!rlang::sym(nom_variable) + 1]
}C’est l’une des raisons pour lesquelles il est préférable d’utiliser data.table lorsqu’on souhaite manipuler fréquemment des données à l’aide de fonctions. Le lecteur intéressé par la question pourra consulter ce post sur le sujet.
Lorsqu’on définit des fonctions pour effectuer des traitements génériques, il est conseillé de privilégier data.table sur dplyr. Une précaution est néanmoins nécessaire pour ne pas modifier les données en entrée de la fonction si l’opérateur := est utilisée. Il est recommandé dans ce cas de créer une copie du dataframe en entrée (data.table::copy(df)) et d’effectuer les traitements sur cette copie.
5.6 Pour en savoir plus
- la documentation officielle de
data.table(en anglais) ; - les vignettes de
data.table:-
Introduction à
data.table(en anglais) ; - Modification par référence (en anglais) ;
-
la foire aux questions de
data.table(en anglais) ;
-
Introduction à
- une cheatsheet sur
data.table(en anglais) ; - une introduction à l’utilisation de l’opérateur
[...](en français) ; - Ce cours complet sur
data.table(en français). - Cette présentation des fonctionnalités du package (en français) ;
- Ce post sur les fonctions utilisant data.table.